介绍
DBSCAN (Density-based spatial clustering of applications with noise) 是一个基于密度的聚类(Cluster)算法:给定空间里的一个点集,该算法能把附近的点分成一组(有很多相邻点的点),并标记出位于低密度区域的噪声点。
原版论文在这里
概念描述及参数
| 标识 | 含义 |
|---|---|
| $D$ | 样本集 |
| $P_i$ | $D$ 中的各样本点 |
| $\epsilon$ (Epsilon) | $P_i$ 的邻域距离阈值 |
| $MinPts$ | $P_i$ 在 $\epsilon$ 范围内样本的数量 |
- 核心点 (Core Points)
- 满足条件,在 $\epsilon$ 范围内,具有 $MinPts$ 个样本的样本点 $P_i$,称作核心点。
- 边缘点 (Border Points)
- 在核心点 $\epsilon$ 范围内,且在样本点 $\epsilon$ 范围内的样本数量小于 $MinPts$,称作边缘点
- 噪声点 (Noise Points/ Outlier)
- 非可达点,即不在核心点的 $\epsilon$ 范围内的样本点,称为噪声点
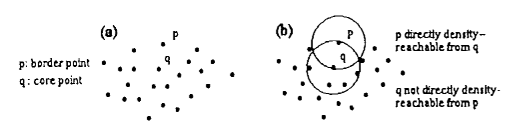
- 直接密度可达 (directly density-reachable)
- 当 $P$ 在除 $P$ 以外的任意样本点 $Q$ 的邻域半径 $\epsilon$ 范围内,且 $Q$ 是一个核心点,则称 $P$ 可由 $Q$ 直接(密度)可达。
- 也就是说,核心点 $P$ 的 $\epsilon$ 范围内除 $P$ 以外的点,都是由 $P$ 直接可达的,同理,非核心点没有直接可达的点。
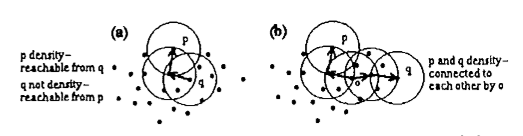
- 密度可达 (density-reachable)
- 对于样本中的点,存在路径 $P_1$,…,$P_n$,使 $P_1$ = $P$,$P_n$ = $Q$,如果该路径中任意一点 $P_{i+1}$ 可以由 $P_{i}$ 直接可达,那么称 $Q$ 是可从 $P$ (密度)可达的。
- 根据该定义,非核心点是可以由其他点可达的,但没有点是由非核心点可达的。
- 参考上图,可达性是非对称的,$P$ 到 $Q$ 可达不代表 $Q$ 到 $P$ 可达。
- 密度相连 (density-connected)
- 对于样本中的点,存在一个点 $O$ 使得 $P$ 和 $Q$ 都是由 $O$ 可达的,则点 $P$ 和点 $Q$ 被称为(密度)相连的,连接性是对称的。定义了连接性后,每个 Cluster 都符合两个性质:
- 同一个 Cluster 中的任意两点都是相连的。
- 如果 $P$ 是由 Cluster 里的点 $Q$ 可达的,那么 $P$ 和 $Q$ 在同一个 Cluster 中。
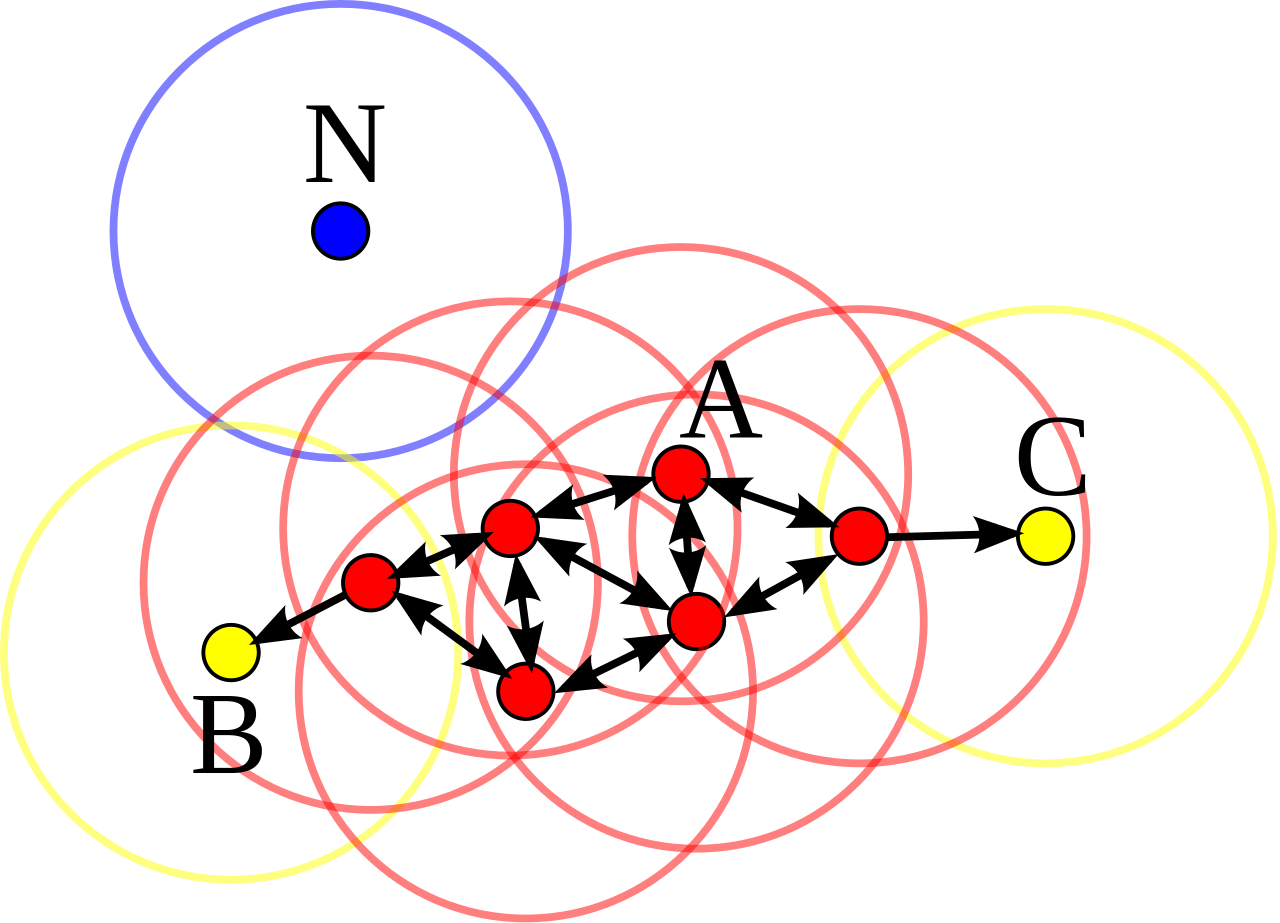
举例
上图中,$MinPts$ = 4,点 A 和其他红色点是核心点,因为它们的 $\epsilon$ 邻域(图中红色圆圈)里包含最少 4 个点(包括自己),由于它们之间相互相可达,它们形成了一个 Cluster。点 B 和点 C 不是核心点,但它们可由 A 经其他核心点可达,作为边缘点加入同一个 Cluster 。点 N 是噪声点,它既不是核心点,又不由其他点可达。
距离函数
这里的距离可以是任意空间内的距离函数,通常使用欧氏距离,在游戏开发中也可能会使用曼哈顿距离。为直观起见,本文中的距离均指的是欧氏距离。
算法
为确定 Cluster, DBSCAN 算法首先从集合里任意一点 $P$ 开始遍历所有密度可达点。然后根据 $\epsilon$ 和 $MinPts$ 判断 $P$ 是核心点还是边缘点。
如果 $P$ 是核心点,该流程会产生一个 Cluster。
如果 $P$ 是边缘点,那么没有任何点是从 $P$ 密度可达的,DBSCAN 将会从样本集中选择下一个点重复该过程。
对于存在多个 Cluster 的情况,算法会根据规则进行合并,这个规则是:
- Cluster 中,对所有 $P$ 的密度可达的点 $Q$ 也属于该 Cluster
- Cluster 中,对所有 $P$ 的密度相连的点 $Q$ 也属于该 Cluster
确定了这些,就明确了 DBSCAN 的算法,简单来说,算法分为下面的步骤:
- 初始化样本集 $D$,以及 $\epsilon$,$MinPts$
- 从任意一点 $P$ 开始查询 $\epsilon$ 邻域内的样本点
- 如果 $\epsilon$ 邻域的点的数量大于等于 $MinPts$,则创建一个 Cluster,否则标记 $P$ 为噪声点(该噪声点也可能位于后续点形成的 Cluster 内)
- 对除点 $P$ 外满足条件的 $\epsilon$ 邻域内的样本点,逐个查询这些点 $\epsilon$ 邻域内的样本点
- 如果这些点 $\epsilon$ 邻域的点的数量大于等于 $MinPts$,便将这些点加入此次创建的 Cluster 中
- 从样本中其他点出发,重复步骤 2 直到遍历完成
论文 4.1 中提供了伪代码:
1
2
3
4
5
6
7
8
9
10
11
12
DBSCAN(SetOfPoints, Eps, MinPts)
// SetOfPoints is UNCLASSIFIED
ClusterId := nextId(NOISE)
FOR i FROM 1 TO SetOfPoints.size DO
Point := SetOfPoints.get(i)
IF Point.ClId = UNCLASSIFIED THEN
IF ExpandCluster(SetOfPoints, Point, ClusterId, Eps, MinPts) THEN
ClusterId := nextId(ClusterId)
END IF
END IF
END FOR
END // DBSCAN
以及创建聚类的 ExpandCluster 函数,经过该函数可以生成一个 Cluster:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
ExpandCluster(SetOfPoints, Point, ClId, Eps,MinPts) : Boolean
//
// regionQuery 函数用于查询并返回以当前点 Point 为中心的 Eps 邻域中的点
// seeds 是包含了从点 Point 出发的所有可达点的集合
//
seeds := SetOfPoints.regionQuery(Point, Eps)
IF seeds.size < MinPts THEN // no core point
//
// 对 Eps 邻域内的样本数量不足 MinPts 个的非核心点(边缘点),由于非核心点没有可达点,因此返回 false。
// 这里虽然标记为 NOISE 但在后续步骤可能会改变该标记
//
SetOfPoint.changeClId(Point, NOISE)
RETURN False
ELSE // all points in seeds are density-reachable from Point
//
// 这里的点是除了初始点 Point 外所有满足 MinPts 和 Eps 的直接密度可达的点,
// 因此先将这些点加入同一个 Cluster 中,并将初始点 Point 删除
//
SetOfpoints.changeClId(seeds, ClId)
seeds.delete(Point)
WHILE seeds <> Empty DO
currentP := seeds.first()
//
// 从该 Cluster 中的其他点出发,查找其他的 Eps 邻域内可达的点
//
result := SetOfpoints.regionQuery(currentP, Eps)
IF result.size >= MinPts THEN
FOR i FROM 1 TO result.size DO
resultP := result.get(i)
//
// 如果可达点暂未划分到其他 Cluster 中(包括此前被标记为 NOISE 的),便将该点加入到该 Cluster 中
// 若此前未归类过,便将该可达点加入可达点的集合 seeds 中
//
IF resultP.ClId IN {UNCLASSIFIED, NOISE} THEN
IF resultP.ClId = UNCLASSIFIED THEN
seeds.append(resultP)
END IF
SetOfPoints.changeClId(resultP, ClId)
END IF // UNCLASSIFIED or NOISE
END FOR
END IF // result.size >= MinPts
//
// 查询完成后从将当前点从点集中删除
//
seeds.delete(currentP)
END WHILE // seeds <> Empty
RETURN True
END IF
END // ExpandCluster
评估
参数
参数的选择向来是个令人头大的问题,在对样本集缺少了解的情况下,很难选出合适的参数。论文中提到了一种叫做 k-distance 的选择 $\epsilon$ 的方法,我将在应用时加以解释。但是即使如此,这个过程也是要不断迭代,最好在数据可视化时进行调整。
假如样本集中的点密度差异较大,也很难产生一个满足所有情况的聚类结果,因为全局的参数 $\epsilon$ 和 $MinPts$ 只有一套。密度变化较大的情况,可以考虑使用 OPTICS 或者 HDBSCAN。
稳定性
DBSCAN 的结果是确定的,对于给定顺序的数据集来说,相同参数下生成的 Clusters 是相同的。然而,当相同数据的顺序不同时,生成的 Clusters 较之另一种顺序会有所不同。
首先,即使不同顺序的数据集的核心点是相同的,Clusters 的标签会取决于数据集中各采样点的顺序。其次,可达点被分配到哪个 Clusters 也是会受到数据顺序的影响的,比如一个边缘采样点位于一个分属于两个不同的 Clusters 的核心点的 $\epsilon$ 邻域内,这时该边缘点被分配到哪个 Cluster 中取决于哪一个 Cluster 先创建。
因此说 DBSCAN 是不稳定的。
效率
算法中遍历了所有的样本点,但实践中算法的时间消耗往往受到 regionQuery 函数的制约。
对于 regionQuery 函数,论文中提到可以使用 R* 树来创建空间索引以提高效率,实践中也可以使用其他的空间划分方法,比如 kd 树。算法期望 $\epsilon$ 邻域范围内的点的数量比起整个样本集的数量是一个较小的值,因此每次查询的时间复杂度为 O(logn)。由于对列表中的每个点的都要进行一次 regionQuery,因此 DBSCAN 的平均的时间复杂度为 O(n * logn)。
当 $\epsilon$ 较大,且 $D$ 数量也比较庞大时,regionQuery 建树的时间消耗会非常大。